
Il genoma più misterioso?
È quello dell’olivo.
 Mentre il sequenziamento del codice genetico della vite è stato realizzato 14 anni fa, quello del melo 10, quello del grano tenero è disponibile da tre anni, i tentativi per realizzare lo stesso obiettivo su Olea europea sono finora falliti o hanno prodotto risultati molto parziali. I 23 cromosomi dell’olivo hanno infatti custodito finora i propri segreti grazie alla dimensione (1,5 miliardi di basi per ciascun aplotipo, il triplo rispetto alla vite), all’elevato grado di eterozigosità, all’indisponibilità di genotipi aploidi e al numero elevato di sequenze lunghe e ripetute (>20%).
Mentre il sequenziamento del codice genetico della vite è stato realizzato 14 anni fa, quello del melo 10, quello del grano tenero è disponibile da tre anni, i tentativi per realizzare lo stesso obiettivo su Olea europea sono finora falliti o hanno prodotto risultati molto parziali. I 23 cromosomi dell’olivo hanno infatti custodito finora i propri segreti grazie alla dimensione (1,5 miliardi di basi per ciascun aplotipo, il triplo rispetto alla vite), all’elevato grado di eterozigosità, all’indisponibilità di genotipi aploidi e al numero elevato di sequenze lunghe e ripetute (>20%).
Ma tutti i “segreti” custoditi nel codice genetico dell’olivo stanno finalmente venendo a galla grazie a Olgenome.
Articolo pubblicato su Terra e Vita 16/2021
Abbonati e accedi all’edicola digitale
La diffusione dei risultati
Un progetto di ricerca ideato e coordinato dal Crea, Centro di Ricerca Olivicoltura, Frutticoltura e Agrumicoltura di Rende (Cs), finanziato dal Mipaaf nell’ambito del Piano Olivicolo Nazionale, i cui risultati sono stati divulgati nel corso di un evento digitale del Crea moderato da Terra e Vita. Realizzato anche grazie al supporto di Iga Technology Services per il sequenziamento e l’assembling del genoma e del Crea Genomica e Bioinformatica per lo sviluppo di una mappa genetica e l’annotazione funzionale del genoma.

«Abbiamo concentrato l’attenzione – spiega Fabrizio Carbone del Crea OFA e coordinatore del progetto - sulla cultivar Leccino, tra le più diffuse in Italia e importante anche per i caratteri di tolleranza a Xylella fastidiosa».
Questi gli obiettivi realizzati da Olgenome:
- sequenziamento e assembling di ciascuno dei due aplotipi presenti nel genoma diploide altamente eterozigote della cv. Leccino (2 miliardi di coppie di basi su 3 miliardi totali);
- identificazione e caratterizzazione dei geni espressi nella cv Leccino per l’annotazione funzionale del genoma (identificando 89mila geni, isoforme escluse);
- realizzazione di un genome browser consultabile dal portale del progetto Olgenome (http://olgenome.crea.gov.it/);
- definizione di un trascrittoma di riferimento della cv Leccino per supportare l’identificazione di geni chiave per l’espressione dei caratteri d’interesse.
Primo passo per il breeding
«Le informazioni genomiche ottenute – afferma Enzo Perri, direttore del Crea Ofa - sono trasversali e utili per tutte le linee di ricerca sull’olivo, da quella strettamente agronomica a quella fitosanitaria o per far fronte agli effetti del climate change. Da quella sulla prima trasformazione a quella organizzativa, incidendo così sulla competitività dell’intero sistema olivicolo».
«I risultati di Olgenome – commenta Stefano Vaccari, direttore generale del Crea – arrivano in un momento decisivo per l’agricoltura europea, contemporaneamente alla pubblicazione dello studio della Commissione Ue che riconosce il ruolo delle biotecnologie di precisione nella realizzazione degli obiettivi del Farm to Fork». «Il settore dell’olio d’oliva italiano – continua- vive un momento molto difficile con stock in aumento e consumi stagnanti. Il Crea ha le risorse umane, le tecnologie e le competenze per sviluppare nuove varietà di olivo o per valorizzare il potenziale anche salutistico delle varietà esistenti».
La conoscenza del genoma è infatti il primo passo necessario per indirizzare i programmi di miglioramento genetico con tecniche convenzionali (incrocio) o avanzate (biotecnologie).
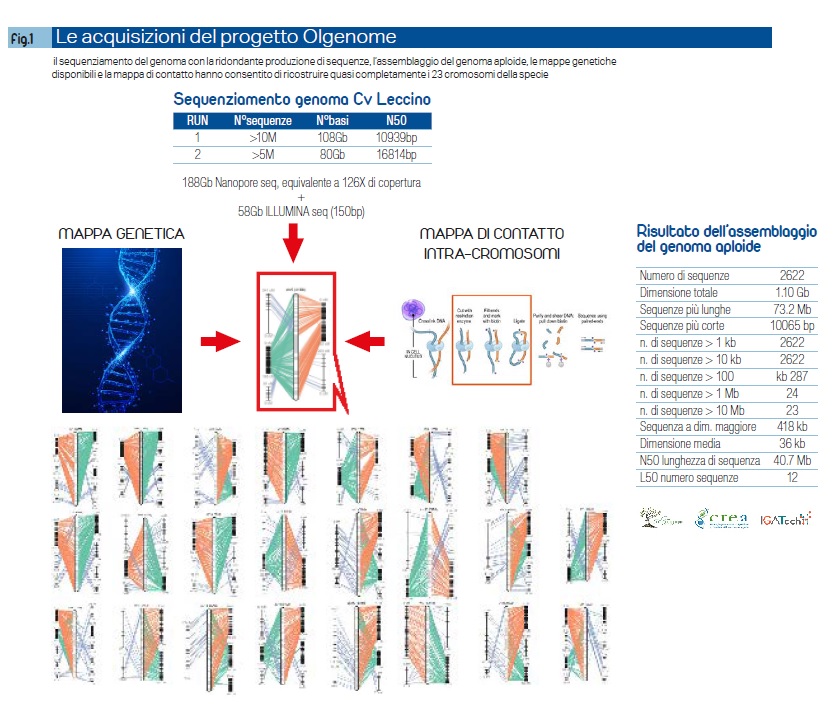
Tab 1. I numeri del sequenziamento genoma Cv Leccino
| RUN | N°sequenze | N°basi | N50 |
| 1 | >10M | 108Gb | 10939bp |
| 2 | >5M | 80Gb | 16814bp |
Un puzzle con troppi pezzi
La collaborazione con Iga Technology Services, spin-off dell’Università di Udine, ha consentito di utilizzare una tecnologia molto evoluta, Oxford Nanopore, che consente di sequenziare anche frammenti lunghi di DNA superando gli scogli che avevano bloccato le precedenti esperienze di sequenziamento.
Hanno tentato, nel recente passato, alcuni gruppi di ricerca spagnoli e turchi. Nel 2016 il sequenziamento del genoma dell’antica cultivar Farga ha consentito di codificare 52mila geni, ma non è stato possibile ancorarli ad una mappa genetica. Nel 2017 un progetto turco spagnolo ha sequenziato il genoma dell’Oleastro (solo 48-50 mila geni comprese le isoforme). Nel 2020 un gruppo spagnolo ha sequenziato la varietà Picual e parzialmente anche altri 40 genotipi per studi evoluzionistici.
«Ma Olgenome – assicura Carbone – con il sequenziamento della cv Leccino, la costruzione della mappa dei 23 cromosomi e l’annotazione funzionale dei geni consente un vero salto di qualità». «Assemblare un genoma – spiega Simone Scalabrin di Iga – è come costruire un puzzle con molti più pezzi del necessario». Una ridondanza cercata e utile perché i frammenti di DNA non combaciano perfettamente.
Nel caso di Olgenome sono state sequenziate più di 100 copie del genoma, 100 tessere per ogni porzione del puzzle. Collocate poi al giusto posto in ogni cromosoma facendo riferimento alle mappe genetiche già disponibili (Leccino e Dolce Agogia) e alla costruzione di una mappa di contatto intra-cromosomi che ha consentito ai ricercatori di orientarsi nonostante l’elevato grado di eterozigosità che caratterizza la specie.
«In questo modo abbiamo potuto ricostruire e mappare 2miliardi di paia di basi (3 miliardi il totale del genoma diploide). Un ottimo risultato perché alcune regioni risultano insequenziabili per la forte colonizzazione di sequenze satelliti che costituiscono fino a un quarto del genoma totale. Sequenze identiche, molto brevi, ripetute una dietro l’altra».
Tab. 2 I principali risultati dell’assemblaggio del genoma aploide
| Numero di sequenze | 2622 |
| Dimensione totale | 1.10 Gb |
| Sequenze più lunghe | 73.2 Mb |
| Sequenze più corte | 10065 bp |
| n. di sequenze > 1 kb | 2622 |
| n. di sequenze > 10 kb | 2622 |
| n. di sequenze > 100 | kb 287 |
| n. di sequenze > 1 Mb | 24 |
| n. di sequenze > 10 Mb | 23 |
| Sequenza a dim. maggiore | 418 kb |
| Dimensione media | 36 kb |
| N50 lunghezza di sequenza | 40.7 Mb |
| L50 numero sequenze | 12 |
Dal genoma ai geni
L’annotazione funzionale è l’attività che consente di passare dal genoma ai geni. Paolo Bagnaresi del Crea GB racconta che il primo passo effettuato nell’ambito del progetto è stato quello di mascherare la componente ripetuta (simple repeats ed elementi trasponibili) che in Leccino rappresentano il 55% del genoma.
«Per identificare i geni – dice – abbiamo utilizzato il software Augustus, basandoci sui dati effettivi di espressione ricavati dalla collezione di 800 milioni di “reads” ricavati dai vari tessuti di olivo ad integrazione dei dati ab initio».
Il risultato è stata la codifica di 88.708 geni, isoforme escluse. Più 109.572 isoforme, che sono piccole varianti di trascrizioni. «Su 89mila geni, 60 mila sono supportati da dati di espressione. I rimanenti possono essere geni di cui non abbiamo identificato le condizioni di espressione oppure pseudo geni». Il matching con geni noti anche in altre specie vegetali ha consentito di identificare geni molto importanti associati all’espressione genica, alla risposta agli stimoli e alle condizioni di stress (10% del genoma totale).
Il trascrittoma di riferimento
L’identificazione e caratterizzazione effettiva dei geni espressi in olivo fa parte dell’attività curata da Amelia Salimonti del Crea Ofa.
«I geni chiave di Leccino possono essere individuati grazie alle attività di trascrittomica, ma le pubblicazioni sono molto limitate e rimane ancora molto da indagare». Per questo nell’ambito di Olgenome si è proceduto alla definizione di un trascrittoma di riferimento della cv Leccino mediante tecnologie di sequenziamento Ngs applicate a campioni di RNA ottenuti da un elevato numero di tessuti e organi differenti, prelevati in diverse fasi fenologiche e in differenti condizioni di stress. In particolare sono state sviluppate due distinte attività:
- trascrittomica e biologia fiorale, per approfondire meccanismi che sottendono all’induzione/ differenziazione fiorale con l’obiettivo di identificare geni target che contribuiscono al complesso network molecolare correlato all’alternanza di produzione;
- trascrittomica e luce continua, per lo studio del processo di fotopercezione.
Quest’ultima attività può dimostrarsi utile a capire meccanismi che possano ad esempio accorciare la fase giovanile anticipando l’entrata in produzione dell’olivo (circa 15 anni).
«Nell’ambito di queste attività- conclude Salimonti – sono stati selezionati alcuni geni con un ruolo chiave in questi processi fisiologici che potrebbero rappresentare nuovi target per le attività di genome editing a supporto dei programmi di miglioramento genetico».

Le opportunità dal DNA alla tavola
Una tavola rotonda virtuale per analizzare l’apporto innovativo che può offrire Olgenome nella risoluzione di problemi cronici dell’olivicoltura nazionale. Ma anche di problemi specifici come la necessità di aumentare la biodiversità, tutelare la tipicità, fare fronte al climate change, progettare una nuova olivicoltura che valorizzi i nostri punti di forza, affrontare emergenze sanitarie come la Xyella.
«Solo il 3% degli impianti italiani – esordisce Stefano Francia di Agia-Cia – ha meno di 15 anni e solo il 5% degli olivicoltori ha meno di 40 anni. Difficile pensare al futuro dell’olivicoltura senza un necessario cambiamento. La biodiversità olivicola è importante perché caratterizza anche il nostro paesaggio, ma per proteggere questa ricchezza servono strumenti nuovi come le New breeding techniques».
«La gamma italiana poggia su 530 cv – evidenzia Verona Barbati di Coldiretti Giovani impresa – e la biodiversità può essere una chiave decisiva anche in ottica commerciale come dimostrano gli oli monovarietali».
«Ma oltre ai futuri possibili bisogna dare risposte anche al presente, rappresentato dall’espansione degli impianti intensivi che si basano su un numero limitato di varietà straniere che nei nostri areali accusano problemi. La conoscenza e la ricerca sono le chiavi per trovare risposte tutte italiane per problemi come questi».
«Se il clima cambia – afferma Francesco Mastrandrea di Anga Confagricolura – devono cambiare le piante e soprattutto l’olivo, che negli ultimi anni sta accusando i problemi maggiori proprio nelle aree vocate. Le nuove biotecnologie possono essere una soluzione, ma occorre essere veloci».
«L’editing può fornire le soluzioni – ribatte Michele Morgante dell’Università di Udine – solo se ha a disposizione un elevato livello di informazioni per individuare i geni su cui intervenire e per questo è importante continuare a sostenere progetti di ricerca post genoma».
«Abbiamo l’obbligo morale – sostiene Luigi Cattivelli del Crea MB –di lasciare più biodiversità ai nostri figli e la strada per farlo è credere nel miglioramento genetico. Nell’agricoltura deve poter coesistere il vecchio con il nuovo. Ma se non pensiamo noi alle nuove varietà, lo faranno i competitor».
«La genomica in Italia è nata dalla ricerca vegetale – commenta Mario Enrico Pè, presidente di Siga- e ora è la chiave per cercare una soluzione anche al problema covid. Olgenome mostra che in Italia ci sono centri di eccellenza e che le tecnologie di evoluzione assistita possono dare risposte adeguate alle esigenze della nostra olivicoltura».
«In Italia oggi – conclude Samanta Zelasco del Crea Ofa- abbiamo due carte vincenti da giocare: la ricchezza della biodiversità delle numerose varietà coltivate che sono uno straordinario serbatoio di geni da cui attingere e ora la mappa codificata del genoma dell’olivo».
«Due ingredienti da cui partire per costruire l’olivicoltura del futuro».
Xylella, la soluzione passa dalla genetica
La soluzione al problema Xylella pasa necessariamente dal miglioramento genetico. Samanta Zelasco del Crea Ofa spiega che un recente lavoro scientifico sulla trascrittomica ha analizzato le differenze tra una varietà tollerante come Leccino rispetto a una suscettibile come Ogliarola salentina individuando i geni sovraespressi nella pianta tollerante se infettata.

«Il problema è che il 40% dei geni non è potuto essere annotato in mancanza di un genoma di riferimento. Ora c’è e il genome browser del progetto Olgenome può consentire di capire i meccanismi coinvolti nella relazione ospite patogeno».



















